Grazie alla tecnologia di sequenziamento ad alta risoluzione e specifici algoritmi di calcolo è possibile analizzare i frammenti del DNA fetale presente nel plasma materno per valutare anomalie a carico dell’intero corredo cromosomico.
Whole Genome Analysis (WGA)
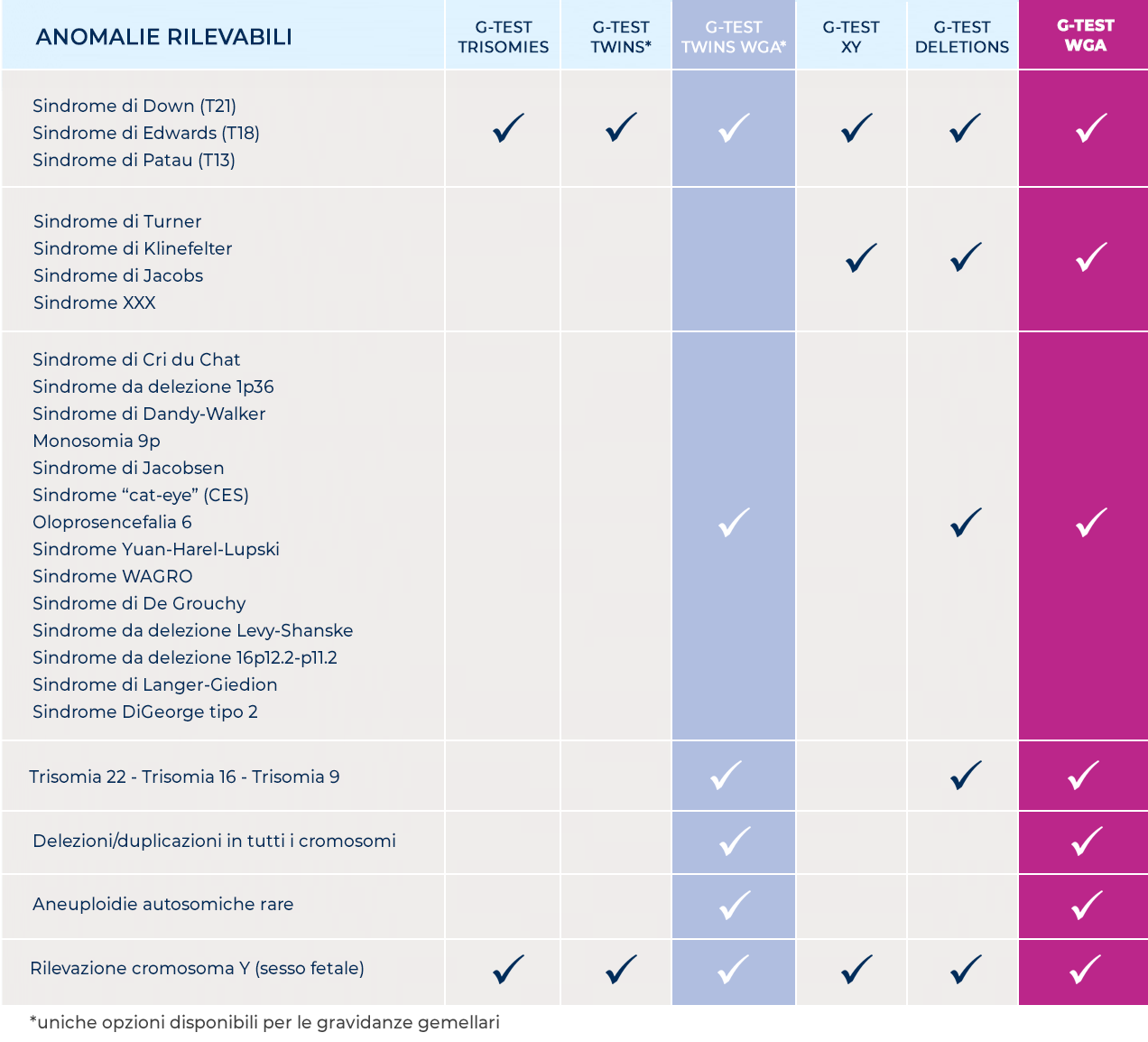
L’analisi dell’intero genoma rende possibile l’ampliamento della ricerca di trisomie, monosomie e delezioni/duplicazioni a tutti i cromosomi. Il G-test prevede un protocollo di analisi certificato CE-IVD basato sull’automazione e sul sequenziamento paired-end ad elevata risoluzione (fino a 60 milioni di reads). Ciò garantisce da una parte, la standardizzazione qualitativa dei flussi di lavoro, dall’altra la possibilità di rilevare trisomie, monosomie e delezioni/duplicazioni, anche di piccolissime dimensioni, a carico dell’intero corredo cromosomico, mantenendo un bassissimo tasso di risultati falsi positivi.
Indicazioni
• Gravidanze singole o gemellari (massimo due feti)
• Gravidanze singole in cui la diagnosi prenatale invasiva è controindicata
• Gravidanze singole o gemellari da fecondazione assistita (omologa e eterologa)
• Gravidanze in cui l’età dei genitori rappresenta un fattore di rischio
• Gravidanze con esito di alto rischio da test prenatali su base statistica
Affidabilità
• Protocollo di analisi interamente certificato CE-IVD
• Sequenziamento del DNA ad elevata risoluzione (fino a 60 milioni di reads)
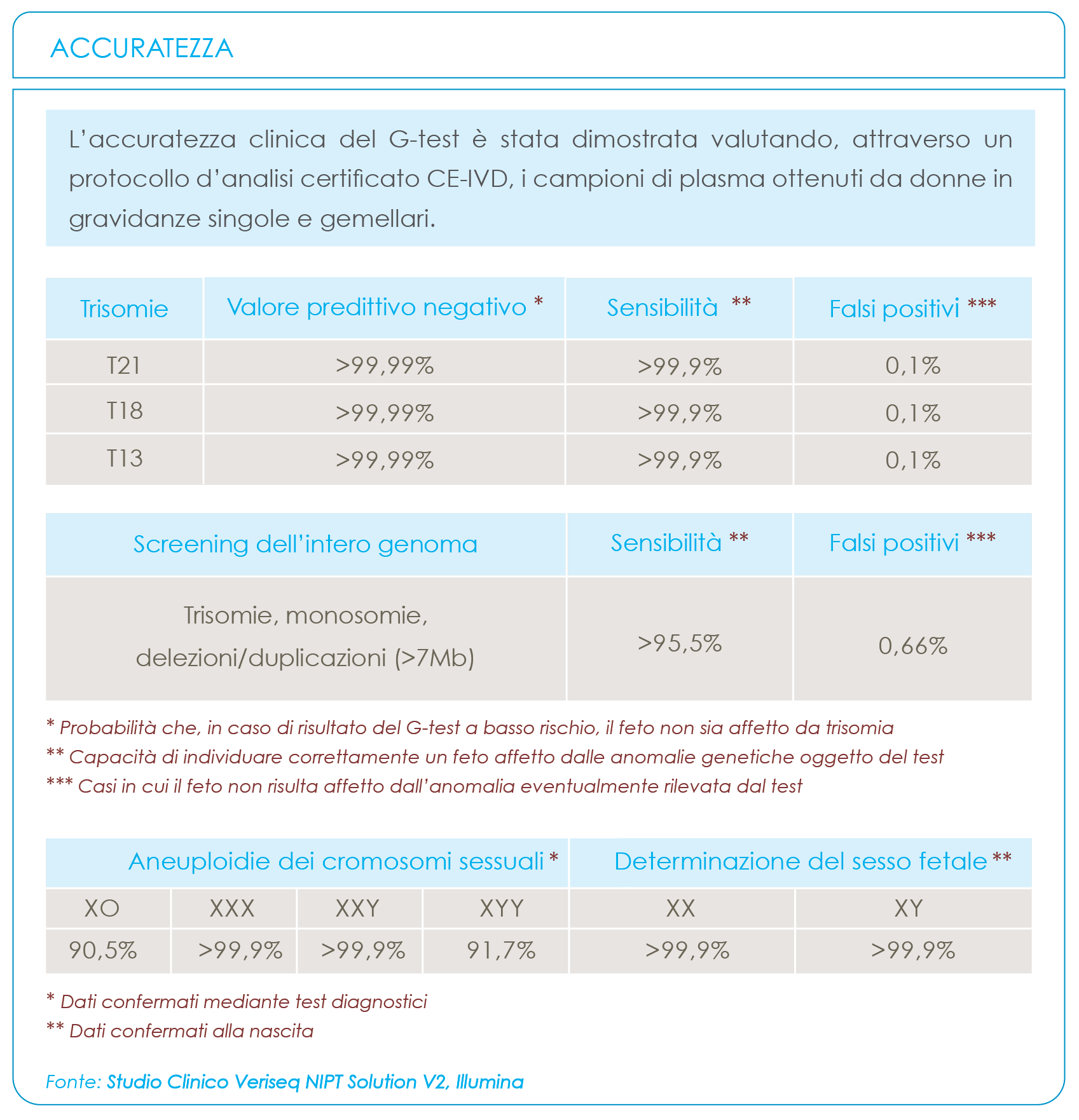
• Sensibilità maggiore del 99,9% per le trisomie 21, 18 e 13
• Falsi positivi per le trisomie 21, 18 e 13 inferiori allo 0,1%
• Sensibilità maggiore del 95,5% per lo screening dell’intero genoma
• Specificità maggiore del 99,3% per qualunque opzione di analisi
• Rilevazione di delezioni/duplicazioni di piccolissime dimensioni
Vantaggi
• Richiede un normale prelievo di sangue, quindi, nessun rischio abortivo
• Si esegue a partire dalla 10a settimana di gestazione
• Ha una elevatissima accuratezza
• Prevede un rimborso* per l’eventuale consulenza genetica
• Prevede un rimborso* per eventuali spese di approfondimento diagnostico
• Risultati in circa 5 giorni lavorativi**, per qualunque opzione di analisi
* Il rimborso è soggetto a limitazioni. Per maggiori dettagli è possibile contattare il numero verde prima di sottoporsi al test
** I tempi si intendono dall’arrivo presso i laboratori e possono variare in funzione delle caratteristiche del campione
Prelievo e trasporto
Il prelievo di sangue periferico materno richiesto per il G-test è di circa 8ml; può essere eseguito a partire dalla 10ᵃ settimana di gestazione utilizzando la provetta certificata CE-IVD fornita nel kit di prelievo e trasporto G-test. Il sangue prelevato viene inserito in un kit specifico per il trasporto di materiale biologico, nel rispetto della normativa UN3373, e inviato ai laboratori di Bioscience Genomics, ubicati presso il Dipartimento di Biologia dell’Università di Roma Tor Vergata, dove vengono svolte tutte le fasi dell’analisi, secondo un rigoroso protocollo certificato CE-IVD.